2013年4月28日作成
2013年9月8日最終更新(文章を読み易くし、図を増やしました)
はじめに
1.距離測定法
2.幾何学的形態測定学
ランドマーク法
楕円フーリエ法
セミランドマーク法
ランドマーク法の具体的な手順
1.写真撮影
2.ランドマーク座標の取得
3.解析
はじめに
生物の形態を定量化する方法は、大きく分けると2つあります。 距離測定法と幾何学的形態測定学(geometric morphometrics)による方法です。 前者は記載学に、後者は進化の研究などによく用いられます。 この小文では、まず両者の特徴を簡単に紹介し、続いて幾何学的形態測定学の手法のひとつであるランドマーク法に的を絞って、写真の撮り方やソフト(tpsDig2とMorphoJ)の使い方を説明します。
1.距離測定法
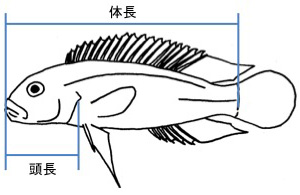
体長や頭長など、形質の長さ、高さ、幅などの距離を測定します。 古くから用いられ、伝統的測定法と呼ばれることもあります。 過去の知見との比較が重要な記載学で威力を発揮します。
利点(幾何学的形態測定学より優れている点)
| ・ | 古い研究の多くがこの方法を使っているので、情報が蓄積されています。 また、それらの情報と自分のデータの比較が容易に行えます。 例えば、文献に「A種は頭長が体長の30%以下、B種では30%以上」と記載されていたとします。 この場合、手元のサンプルで頭長が28%だったらA種に同定できます。 |
| ・ | 記載から形や大きさをイメージし易いです。 例えば、「体長が4センチ、頭長が1センチの魚」と記載されていれば、「だいたいメダカくらいかなぁ・・・」と想像がつきます。 |
| ・ | アロメトリー式を当てはめることが出来るため、形質のプロポーション(割合)が成長によってどのように変化するかを評価し易いです。 例えば、頭長 = a 体長bとしたとき、b < 1であれば体長が大きくなるに従って頭が相対的に小さくなり、b > 1なら相対的に大きくなることを表しています。 b = 1なら、体長が変化しても頭長の割合が一定であることを表しています(イソメトリー、頭長/体長 = a)。 |
欠点(幾何学的形態測定学より劣っている点)
| ・ | 角度や輪郭の形を数値化するのには向いていません。 例えば、口がどのくらい上を向いているか(角度)や、背中の輪郭がどのくらい丸みを帯びているかをこの方法で数値化するのは難しいです。 |
2.幾何学的形態測定学
比較的新しい方法で、google scholarで検索すると、この方法を用いた論文は1990年代後半くらいから増え始めたようです。農作物の形の解析や、形態の進化の研究などに応用されています。通常は、シェイプ(かたち)とサイズ(大きさ)を分離して、独立に解析します。ランドマーク法、楕円フーリエ法、セミランドマーク法がよく使われます。
利点(距離測定法より優れている点)
| ・ | シェイプを数値化し、また逆にその数値からシェイプを視覚的に再現することができます。 |
| ・ | ランドマーク法では角度(口の向きなど)を、また楕円フーリエ法とセミランドマーク法では輪郭の形(背中の輪郭の曲がり具合など)を解析するのに優れています。 |
欠点(距離測定法より劣っている点)
| ・ | 過去の研究との整合性をとるのが難しいです。 多くの場合、最終的な出力は主成分得点(もしくはrelative warp scores)ですが、研究によって意味が異なります。 つまり、異なるサンプルを用いた研究Aと研究Bでは、同じ主成分(例えば第1主成分)において主成分得点が同じであっても、シェイプが同じという訳ではありません。 |
| ・ | 形や大きさを記載する「記載学」には向いていません。 |
| ・ | アロメトリー式を当てはめることが出来ません。 しかし、サイズ(セントロイドサイズなど)と相関のある主成分があれば、サイズに沿った形態の変化を視覚的に再現することは可能です。 |
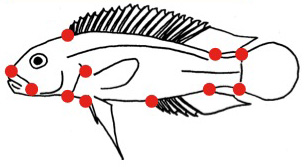
ランドマーク法
「吻端」や「背鰭基底前端」などにランドマークを置いて、その座標をもとに解析します。詳しくは下で説明します。
楕円フーリエ法
輪郭を数値化し、その数値から輪郭を滑らかな曲線として再現できます。 解析には標準化された楕円フーリエ係数を用いますが、同じ係数でもサンプル間で相同と仮定するのが難しいです(サンプル間で相同性のないデータを解析しても良いかについては、議論の余地があります)。 輪郭は閉じている必要があります。

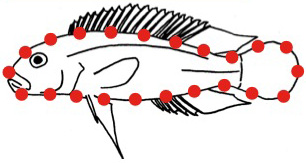
セミランドマーク法
輪郭にセミランドマークを置いて解析します。上述のランドマーク法では吻端など特定の場所(サンプル間で相同な場所)にランドマークを設定しますが、セミランドマーク法では特定の場所ではなく、例えば輪郭上に「等間隔」に設定します。このため、セミランドマークがサンプル間で相同と見なすことは難しいです(楕円フーリエ法と同様に、相同性のないデータの解析には議論の余地があります)。輪郭は閉じていなくても解析できます。
以上の4つの方法のまとめです。
|
シェイプを 視覚的に 再現できる |
輪郭を 解析 できる |
曲線を 再現 できる |
データに 相同性が ある |
他研究と 整合性が ある |
アロメト リー式を 使える |
|
| 距離測定法 | × | × | × | ◯ | ◯ | ◯ |
| GMのランドマーク法 | ◯ | × | × | ◯ | × | × |
| GMの楕円フーリエ法 | ◯ | ◯* | ◯ | × | × | × |
| GMのセミランドマーク法 | ◯ | ◯ | × | × | × | × |
*輪郭は閉じている必要があります。
注)この表は簡潔にまとめたもので、例外もあります。
ランドマーク法の具体的な手順
ランドマーク法では、まず1)サンプルの写真を撮り、2)写真からランドマークの座標を取得して、3)座標を統計的に解析します。 写真撮影の方法は対象とする生物によって異なると思いますが、今回は魚を例に説明します。
1.写真撮影
必要なもの
サンプルとなる魚、発泡スチロール板、虫ピン(10本くらい)、デジカメ、写真台、スケール(定規)
手順
| ・ | 写真台にデジカメと発砲スチロール板を設置して、魚をセットします。このとき、ランドマークの位置が分かり易いように工夫します。例えば、下顎関節など写真から位置が分かり難いランドマークは、虫ピンを刺して、その場所を明示しておきます(下の写真の1)。鰓蓋にランドマークをとるときは、鰓蓋の輪郭がはっきりするように、目立つ色の紙片を挟んでおきます(同2)。鰭の基部にランドマークをとるときは、鰭を虫ピンで立てておきます(同3)。 |
| ・ | 撮影します。忘れずにスケールを写し込んで下さい。後で写真を加工するときに必要となります。 |
ヒント
| ・ | 全てのサンプルで、同じ姿勢となるようにします。例えば、サンプルによって体の反り具合が異なれば、後の解析に影響を与えてしまいます。このため、最初のサンプルの写真を基準とし、二個体目以降は、最初の写真を見ながら慎重に姿勢を調整して撮影します。しかし、ホルマリンで固定されたサンプルでは、姿勢を変えるのが難しいです。生のサンプルを用いるのがベターです。 |
| ・ | 写真の歪みを少なくするため、やや離れたところからズームで撮るのが良いでしょう。 |
2.ランドマーク座標の取得
必要なもの
上で撮った写真、コンピューター、tpsDig2(フリーソフト、Windows用のみ、http://life.bio.sunysb.edu/morph/)、Photoshop
手順
| ・ | 座標を取得する前に、写真の縮尺を揃えます。方法は、解像度を調節して、写し込んだスケールの長さを全ての写真で同じにします。ここではPhotoshop CS3を使った方法を紹介します。なお、写真の縮尺がもともと全て同じ場合(同じズーム率で、レンズと魚の距離が一定の条件で撮影した場合)、この操作は必要ありません。
|
||||||||||||||||||
| ・ | 次に、tpsDig2で座標を取得します。まず、適当なソフトで下のような入力ファイルを作り、拡張子を.TPSにしてtext形式で保存します(例えばhogehoge.TPS)。保存場所は、写真と同じフォルダにします。
| ||||||||||||||||||
| ・ | tpsDig2を開きます。 | ||||||||||||||||||
| ・ | メニューの「File」(下の図の1)から「Input source」「File」を開き、入力ファイルhogehoge.TPSを開きます。すると写真も自動的に読み込まれます。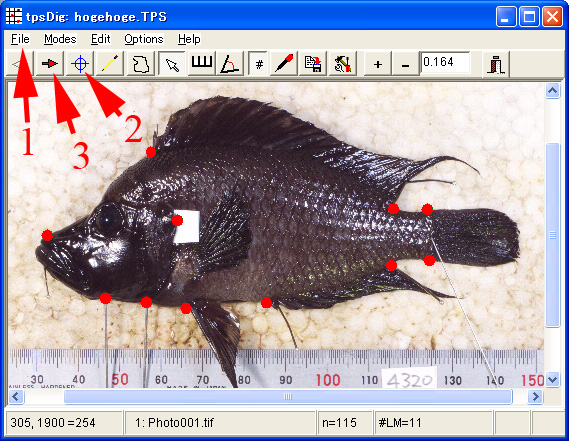 | ||||||||||||||||||
| ・ | パネルの上部にあるボタンのうち、○と十の重なったような絵のボタン(上の図の2)を押します。すると、ランドマークを入力できるようになります。写真上でクリックしてランドマークを決めて行きます。(注意:ランドマークの番号は、全ての写真で相同となるようにして下さい。例えば、吻端は必ず番号1のランドマークとする、などです)。 | ||||||||||||||||||
| ・ | 次の写真に進むには、右向き矢印の絵のボタン(同3)を押します。 | ||||||||||||||||||
| ・ | 全ての写真でランドマークを決め終えたら、メニューの「File」(同1)から「Save data...」で保存します。下のように、入力ファイルhogehoge.TPSにランドマークの座標が書き込まれているはずです。
|
3.解析
必要なもの
コンピューター、tpsDig2で作った入力ファイル、MorphoJ(フリーソフト、Windows, Macなど、さまざまなプラットフォーム用がある、http://www.flywings.org.uk/MorphoJ_page.htm)
少し説明
上で写真から座標を取得しましたが、そのもととなる写真では、サンプルによって中央からズレた位置に写っていたり、また時計周り/反時計回りに傾いていたりします。 このため、取得した座標からその影響を排除する必要があります。 それを行うのがプロクラステス解析で、縦横方向の位置と、傾き具合を揃えます。 またこの解析では、併せて大きさ(サイズ)と形(シェイプ)を分離します(このサイズをセントロイドサイズとよびます)。 つまりプロクラステス解析では、変位(縦横の移動)、回転、スケーリングを行って座標を整え、サンプル間で比較できるようにします。 なおプロクラステス解析の理論的背景については良書があるので、そちらをご覧ください(例えば、三中信宏 1997.生物系統学,東京大学出版会;三中信宏 1999.形態測定学.In 棚部一成,森啓(編)古生物の形態と解析,朝倉書店)。
次に、プロクラステス解析で補正したシェイプ(座標)を主成分分析で解析し、2n - 4次元の主成分に分解します。nはランドマークの数です。次元数が座標の数2nより4少ないのは、プロクラステス解析で自由度が4減少したためです(変位で2、回転で1、スケーリングで1減少)。 この主成分を使って、さらにさまざまな解析ができます。 例えば、ある主成分とある変数(体サイズ、地理情報、生態情報など)が相関する場合、その主成分の表すシェイプと変数の関係が示唆されます。 また、例えば群間(雌雄間や地域集団間など)でシェイプに違いがあるかを検定する場合、解析した個体を群に分けて、主成分得点を使って多変量分散分析(MANOVA)を行うことができます。サイズを考慮したい場合は、Centroid SizeもしくはLog Centroid Sizeを共変量として多変量共分散分析(MANCOVA)をしても良いでしょう。
MorphoJでは、上述のプロクラステス解析と主成分分析を行い、得られた主成分ごとにシェイプを視覚的に再現することができます。下にMorphoJ (Mac版) の使い方を説明します。なお、研究によってはシェイプをアフィン変換による要素と非アフィン変換による要素に分けてどちらか一方だけを解析することがあります。しかしMorphoJでは、このような解析はサポートされていません。両者を分けた解析は、tpsに含まれるtpsrelwでできます。
手順1:ファイルを開く
| ・ | MorphoJを立ち上げます。 |
| ・ | ウィンドウ上部メニューの「File」(下の図の1)から「Create New Dataset ...」を選択し、新たなウィンドウを開きます。 |
| ・ | 「File Type」の「TPS」(同2)をチェックします。 |
| ・ | 先ほどtpsDig2で作った入力ファイルhogehoge.TPSを選択(同3)して「Create Dataset」(同4)を押します。これで、TPSファイルが読み込まれます。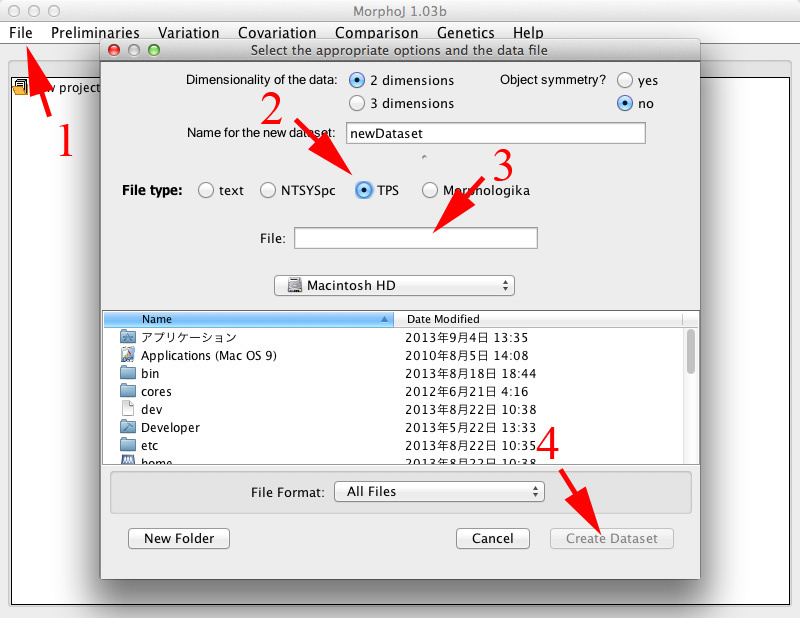 |
手順2:解析
| ・ | ウィンドウ上部メニューの下にボタンが4つあります。そのうち左端の「Project Tree」(下の図の1)を選択します。 |
| ・ | ウィンドウ中の「newDataset」(同2)を選択します。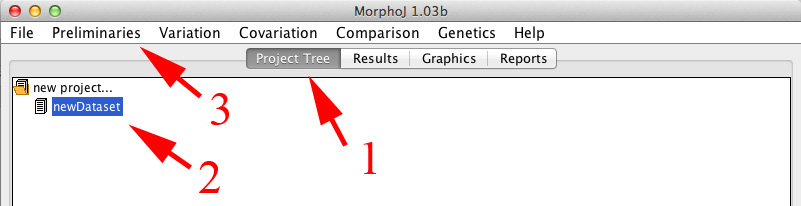 |
| ・ | ウィンドウ上部メニューの「Preliminaries」(上の図の3)から「New Procrustes Fit ...」を選択すると、新たなウィンドウが開きます。その中の「Perform Procrustes Fit」を押すと、プロクラステス解析が行われます。ウィンドウは「Graphics」に自動的に変わり、補正後のランドマークが表示されます(下の図)。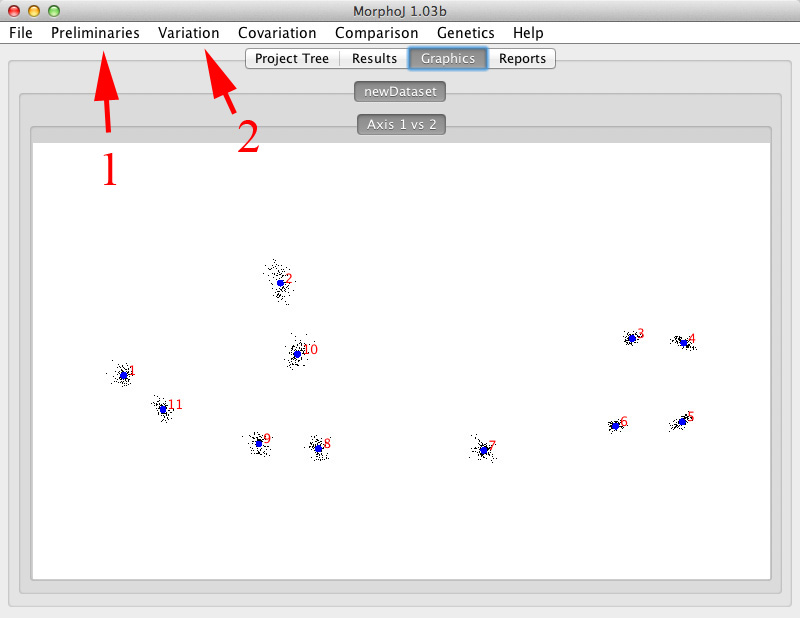 |
| ・ | ウィンドウ上部メニューの「Preliminaries」(上の図の1)から「Generate Covariance Matrix ...」を選択すると、新たなウィンドウが開きます。その中の「Execute」を押すと、分散共分散行列が作られます。 |
| ・ | ウィンドウ上部メニューの「Variation」(同2)から「Principal Component Analysis」を実行すると、第1主成分の軸に沿ったシェイプの変化の図が表示されます(下の図)。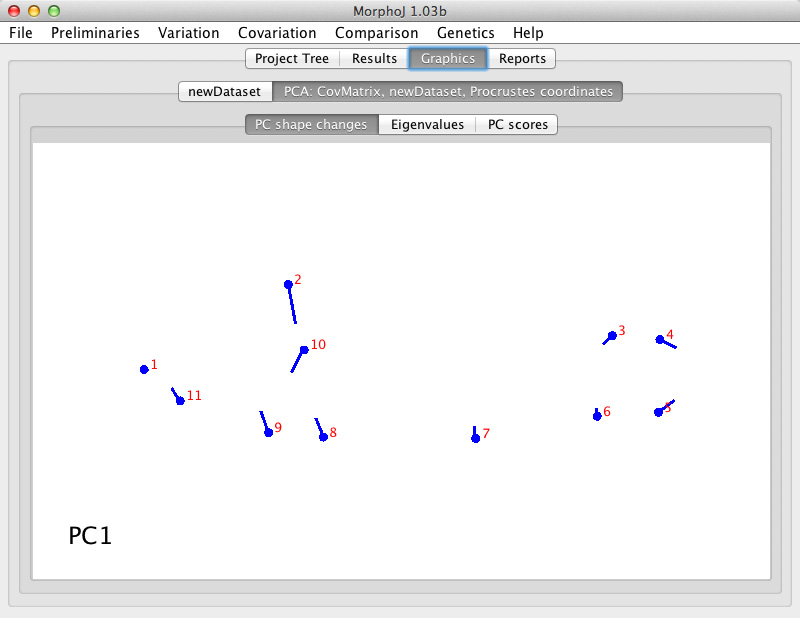 |
| ・ | ウィンドウ上で右クリック(もしくはcontrol + クリック)をするとメニューが出ます(下の図)。このメニューから、さまざまなことができます。例えば、歪んだグリッドの図(下の下の図)は、「Change the Type of Graph ...」から「Transformation Grid」を選びます。また、同メニューの「Choose PC to Display ...」で主成分の軸を変えたり、「Set Scale Factor ...」でscale factorの値を調節することによって、シェイプがどのように変化するかを視覚的に確認できます。図を保存するには、「Export Graph to File ...」を選択します。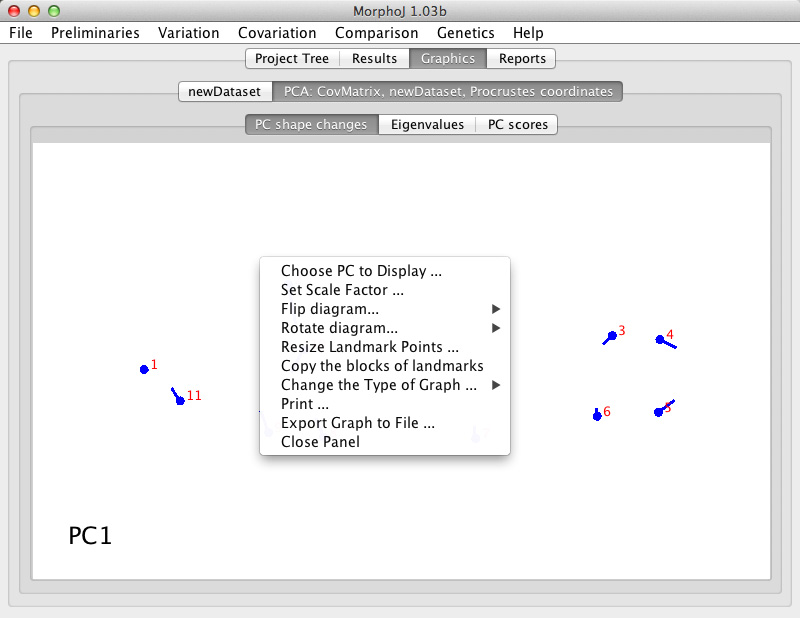 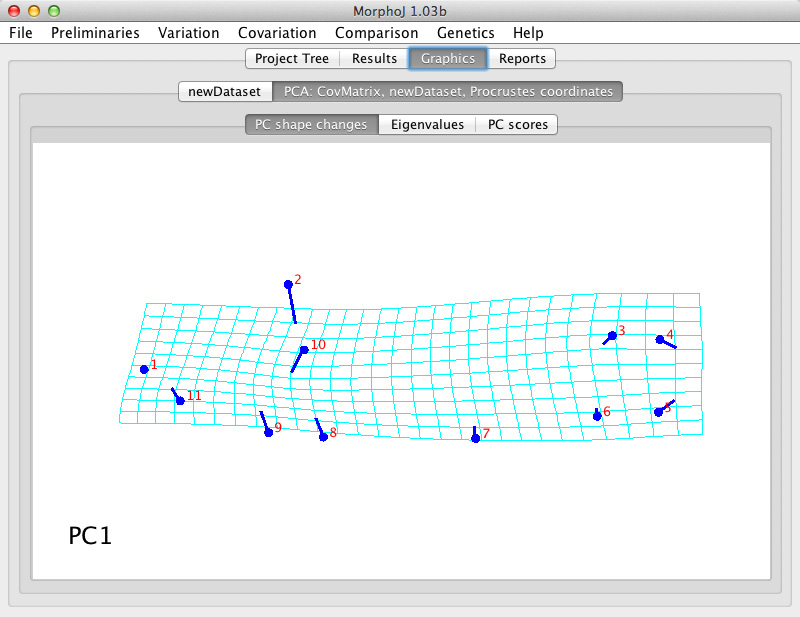 |
手順3:生データ、セントロイドサイズ、プロクラステス座標を保存する
| ・ | ウィンドウ上部にある「Project Tree」ボタン(下の図の1)を押して画面を切り替えます。 |
| ・ | 「newDataset」(同2)を選択します。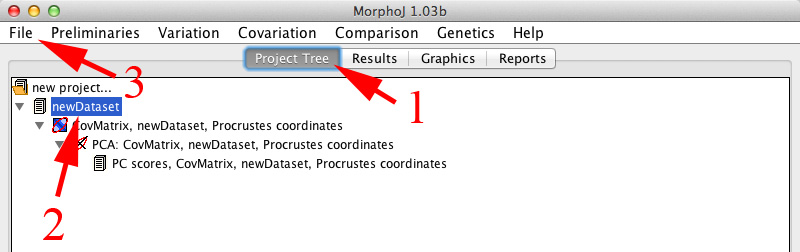 |
| ・ | ウィンドウ上部メニューの「File」(上の図の3)から「Export Dataset ...」を選択すると、新しいウィンドウが開きます。保存したい項目を選択します(下の図の1)。Raw Coordinatesは元の座標、Centroid sizeはサイズを表す数値、Procrustes coordinatesはプロクラステス解析で補正した後の座標です。ファイル名と保存する場所(同2)を指定して「Save」(同3)を押します。なお、保存されたファイルの中で、ランドマークnのx座標とy座標は、それぞれ2n - 1のRawCoord(またはProcCoord)と2nのRawCoord(またはProcCoord)と表示されます。例えばランドマーク1のx座標とy座標はそれぞれRawCoord1とRawCoord2といった具合です。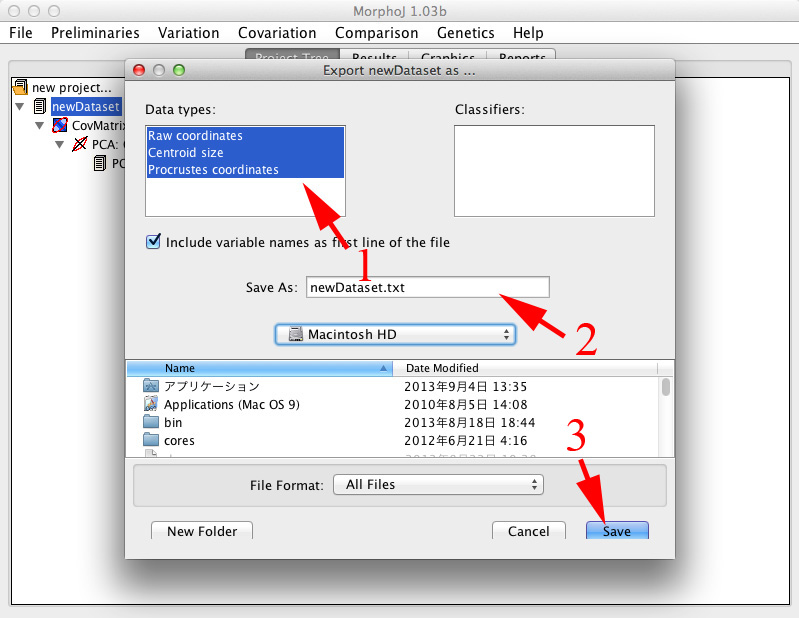 |
手順4:主成分分析の結果を保存する
| ・ | ウィンドウ上部にある「Project Tree」ボタン(下の図の1)を押して画面を切り替えます。 |
| ・ | 「PC scores, CovMatrix, newDataset, Procrustes coordinates」(同2)を選択します。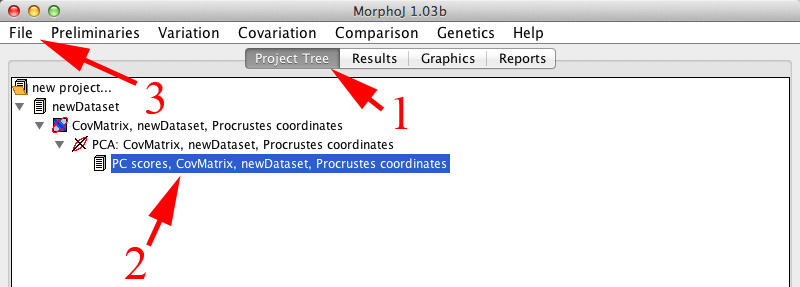 |
| ・ | ウィンドウ上部の「File」(上の図の3)から「Export Dataset ...」を選択して新しいウィンドウを開きます。 |
| ・ | 「PC scores」(下の図の1)を選択して、ファイル名と保存場所(同2)を指定して「Save」(同3)を押します。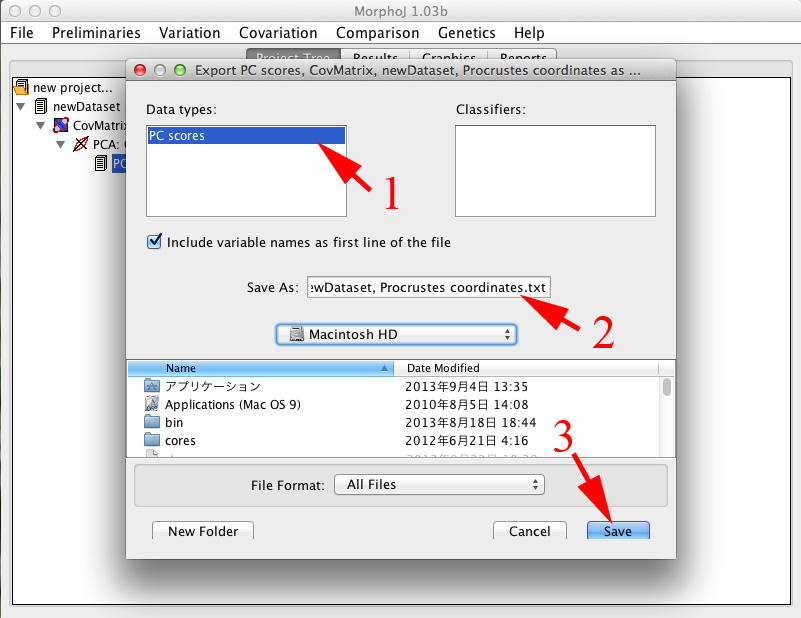 |
手順5:固有値の見方
| ・ | それぞれの主成分が全体の分散のうちどのくらいを説明しているか(寄与率)は、固有値(Eigenvalue)を見ます。ウィンドウ上部のボタンで「Graphics」(下の図の1)を選択し、その下にあるボタンで「PCA: CovMatrix, newDataset, Procrustes coordinates」(同2)を選択し、さらにその下にあるボタンで「Eigenvalues」(同3)を選択します。すると、固有値の割合の棒グラフが表示されます。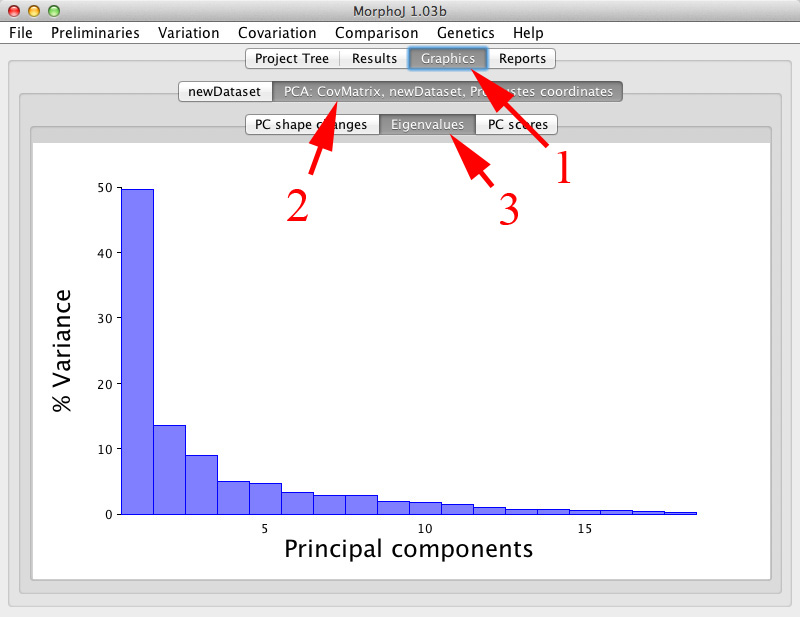 |
| ・ | 固有値の具体的な数値を取得するには、ウィンドウ上のボタンで「Results」(下の図の1)を選びます。主成分分析の結果の中に、固有値が記載されています(同2)。しかし、固有値をファイルに書き出すことが出来ないようです。私は、この画面上の値をエクセルなどにコピペして使っています。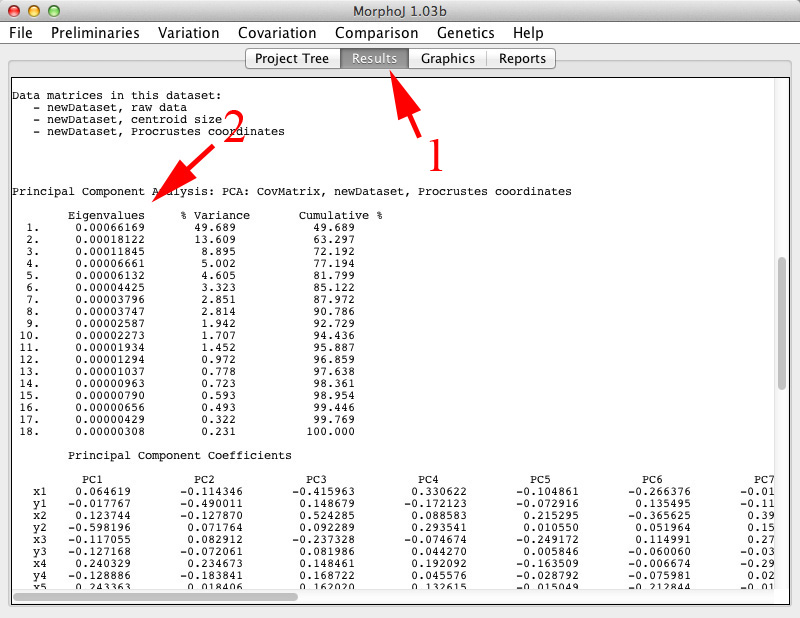 |
ヒント
| ・ | MorphoJには他にも、さまざまなオプションが用意されています。ここでは説明しませんが、いろいろと試してみて下さい。 |
| ・ | 上でも書きましたが、主成分分析の次元数は、ランドマークの数をnとした場合、2n - 4です。これが観察した個体数より多いと、その多い分の主成分には分散がなくなってしまいます(つまり寄与率0)。また、自由度が少ないと、統計上の問題が生じることが予想されます(例えば検出力が下がったり、極端な場合は主成分分析に続く解析自体が出来なくなるなど)。十分な自由度を確保するため、サンプルサイズを大きくすると共に、ランドマークは少なく、しかし調べたいことは表されるように、要領よく設定することが重要と考えます。 |
ホームに戻る